| identific | cha_prestacion | neumococo | Triple viral - SRP | Hepatitis A pediátrica | Varicela | Influenza | |
|---|---|---|---|---|---|---|---|
| 1241443577 | 19/04/2024 | ? | ? | ? | ? | Primera dosis | |
| 1241443577 | 09/08/2024 | Primer Refuerzo | Primera dosis | Única | Primera dosis | Segunda dosis | |
| identific | cha_prestacion | neumococo | Triple viral - SRP | Hepatitis A pediátrica | Varicela | Influenza | Influenza2 |
| 1241443577 | 09/08/2024 | 09/08/2024 | 09/08/2024 | 09/08/2024 | 09/08/2024 | 09/08/2024 | 19/04/2024 |
Hello @migueholguin
Voy a traducir mi respuesta usando Google Translate.
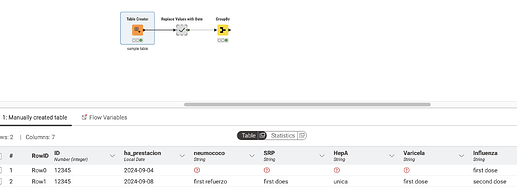
El primer paso que haré es copiar tu entrada. A continuación, he mostrado la misma configuración de tabla que has descrito:
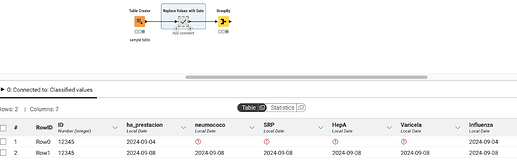
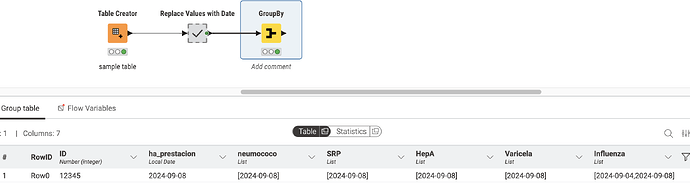
Entonces, para llevar esa tabla a la otra, primero necesitamos reemplazar todos los valores con su fecha correspondiente en la fila. Podemos hacer esto con una serie de nodos Rule Engine donde simplemente buscamos ver si el valor no está ausente. Si el valor no está ausente, lo reemplazamos con la fecha en esa fila. Si tenemos uno para cada columna que necesitamos reemplazar, obtendremos la salida a continuación:
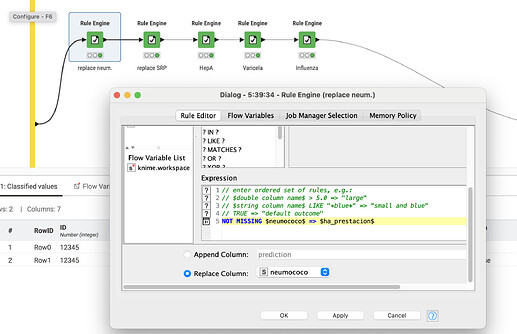
Dentro del metanode, verás cómo hice esto:
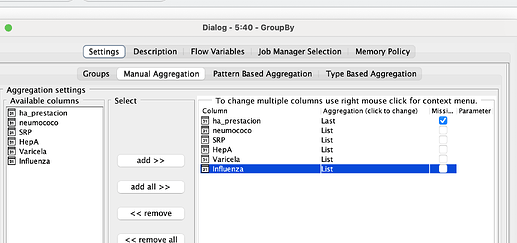
Simplemente reviso cada una de las columnas allí y, como mencioné antes, la reemplazo con la fecha si no está ausente. Al final, uso un group by para agrupar por ‘ID’ o ‘identific’ en tu caso. Ahora, para mi ejemplo, simplemente agregué las otras columnas en una lista y omití los valores faltantes, por lo que no necesitas agregar la columna extra. Si necesitas la columna extra para ‘Influenza2’, probablemente puedas dividir la lista para tener un valor por columna. Creo que la lista es la mejor opción.
A continuación, puedes ver la configuración para la agregación (elegí agrupar solo por ID).
Me aseguro de omitir los valores faltantes o la salida se verá como = [?, 2024-09-08] para las columnas con valores faltantes.
Aquí tienes el resultado:
Aquí tienes el flujo de trabajo a continuación:
Testing.knwf (82.1 KB)
Hope this helps,
TL
Muchas gracias, eres muy amable. lo que me faltaria es identificar el nombre de la dosis, es decir para la influenza debe salir influenza dosis 1 e influenza dosis 2… no se si me hice entender
| ID | ha_prestacion | neumococo | SRP | HepA | Varicela | Influenza_primera_dosis | Influenza_segunda_dosis |
|---|---|---|---|---|---|---|---|
| 12345 | 08/09/2024 | 08/09/2024 | 08/09/2024 | 08/09/2024 | 08/09/2024 | 04/09/2024 | 08/09/2024 |
| algo como asi |
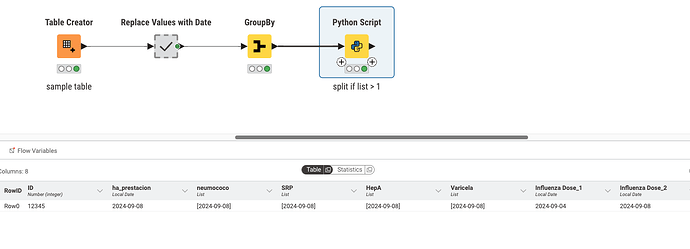
Aquí tienes el flujo de trabajo a continuación; lo he adaptado para dividir la lista en otra columna si hay más de un valor en ella.
Testing.knwf (85.6 KB)
Entonces, no es tan simple hacerlo con nodos, ya que tendrás que renombrar manualmente si lo divides usando uno de los nodos de división de KNIME, pero fui adelante e hice un script de Python que lo hará dinámicamente por ti y lo renombrará. También eliminará automáticamente la columna anterior para que no haya duplicados. Ve el resultado a continuación:
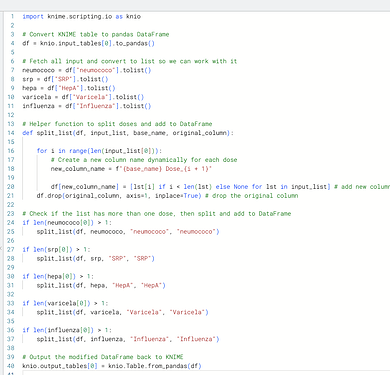
Usé el script de Python para hacer esto y puedo cubrir brevemente el código dentro.
Básicamente, simplemente convierto cada una de esas listas que ves para que sean manejables en Python, luego tengo una función auxiliar, split_list, que dividirá la lista y agregará dinámicamente el nombre, por lo que si tienes 2, 3, 4… dosis, las nombrará adecuadamente.
Después de esto, solo tengo declaraciones if que verifican si la lista tiene más de un elemento, porque no queremos dividirla si no es mayor que uno. Simplemente pasamos la lista, el nombre y el nombre de la columna original a esa función auxiliar que mencioné.
¡No dudes en hacer cualquier pregunta en el foro si necesitas ayuda!
Hope this helps,
TL
Si deseas cambiarlo de inglés a español
Por favor, solo cambia esto en el script de Python.
![]()
Cambias Dose → Dosis
Además, probablemente necesitarás ajustar los nombres de las columnas para que se adapten a tu entrada.
muchas gracias, eres top
This topic was automatically closed 7 days after the last reply. New replies are no longer allowed.